Дизайн трансляции и коррекции: ферменты «двойного сита»
Автор: Др. Джонатан Сарфати (англ. Jonathan Sarfati)
Источник: creation.com
Перевод: Елена Бондаренко
Редактура: Владимир Силенок, Екатерина Савченко
Все живые организмы содержат буквально энциклопедическое количество сложной, специфической информации. Для хранения этой информации у живых существ есть, безусловно, самая компактная система хранения и поиска информации из известных: система белок-нуклеиновые кислоты (белок-ДНК-РНК). Основной план или рецепт закодирован на огромных молекулах ДНК (дезоксирибонуклеиновая кислота).[1] Кодон — последовательность трёх из четырех возможных нуклеотидов, или «букв ДНК», кодирует одну из двадцати аминокислот (или белковых букв). Ген – это последовательность нуклеотидов, которая кодирует один белок, или одну субъединицу многокомпонентного белка. Даже самый маленький известный геном среди свободноживущих организмов — Mycoplasma genitalium – содержит 482 гена, состоящих из 580000 нуклеотидов.[2] (Прим. – статья от 1999 года, а в двухтысячных годах были обнаружены несколько бактерий с ещё более коротким геномом).
Расшифровка (трансляция) генетической информации требует многих компонентов, включая сложную систему репарации для исправления ошибок и повреждений ДНК. Но известный философ науки сэр Карл Поппер (1902 – 1994), обращал внимание на то, что:
«…механизм, при помощи которого клетка (как минимум, не примитивная клетка, а мы только такие и знаем) осуществляет трансляцию ДНК, состоит как минимум из пятидесяти макромолекулярных компонентов, которые сами закодированы в ДНК. Так что трансляция генетического кода невозможна без продуктов трансляции. Получается замкнутый круг; по-настоящему порочный круг для любых попыток сформировать модель или теорию происхождения генетического кода».[3]
Очевидный вывод – механизм трансляции ДНК должен был быть полностью функциональным с самого начала, иначе жизнь бы просто не существовала.
Молекулы трансляции
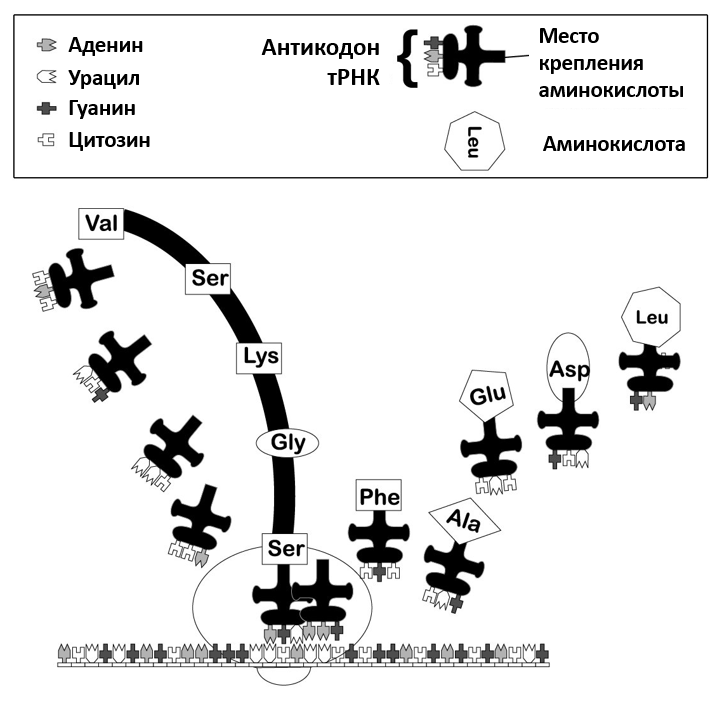
Рисунок 1. Процесс трансляции на мРНК. Рибосома двигается вдоль нити мРНК, наращивая полипептидную цепь. (согласно Jorde, et al.[32]).
Один из многих типов молекул, необходимых для процесса трансляции — это молекулы транспортной РНК – тРНК. Это молекулы, которые соединяют определенную аминокислоту с определенным кодоном. Они состоят из около 80 нуклеотидов, из которых, последовательность трех из которых называется антикодон. Антикодон связывается строго с соответствующим ему кодоном на матричной РНК (мРНК), который, в свою очередь, транслирует определённый код из ДНК. Таким образом, тРНК могут переносить нужные аминокислоты в нужное место в нарастающей пептидной цепи, как это закодировано в мРНК.[4] Кроме того, аминокислота связывается с тРНК таким образом, что становится активированной (то есть с высоким химическим потенциалом) – это необходимо, чтобы образовать пептидную связь с соседней аминокислотой в строящемся полипептиде. Свободные аминокислоты почти не имеют тенденции к самостоятельному образованию полипептидов; скорее, имеет место обратная тенденция.[5]
Согласно законам химии, существуют огромные проблемы с любым эволюционным объяснением того, как нуклеиновые кислоты возникли в гипотетическом первобытном бульоне.[6],[7] Но, даже если мы предположим, что у нас самопроизвольно возникла РНК, есть ещё большое препятствие в том, чтобы правильная аминокислота присоединилась к правильному антикодону естественным образом. Если цепочка аминокислот окажется неправильной, механизм трансляции расшифрует неправильное сообщение, или вообще ничего не расшифрует, то есть организм не сможет синтезировать жизненно важные ферменты. Между прочим, нет никаких химических предпосылок, чтобы какой-нибудь специфический антикодон связывался с какой-нибудь специфической аминокислотой. Ведь они расположены на противоположных сторонах молекулы тРНК, что исключает любые химические взаимодействия. Опять же, всё должно быть полностью функциональным с самого начала.
Синтез тРНК
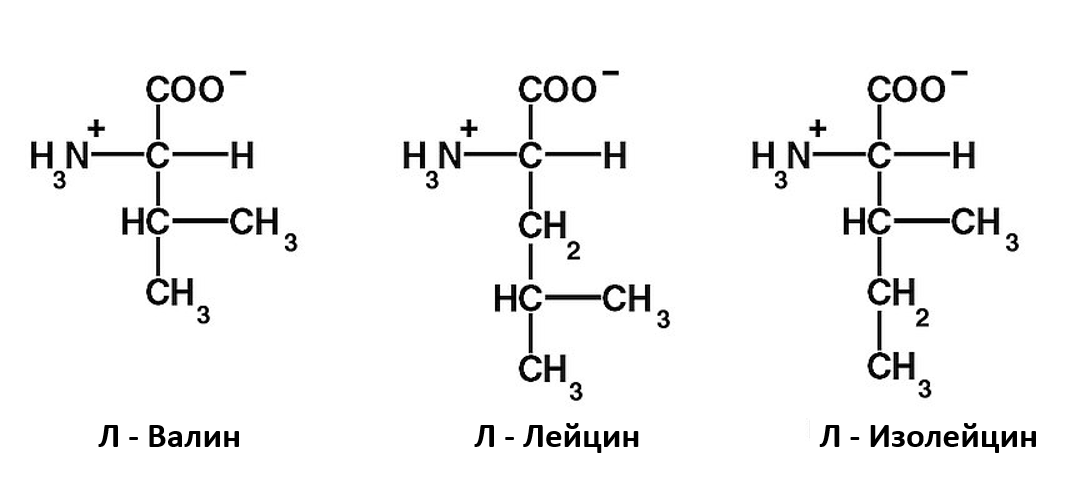
Рисунок 2. Три аминокислоты
Живые организмы не полагаются (да и не могли бы) на случайные химические реакции, чтобы синтезировать тРНК. Напротив, правильная аминокислота активируется и связывается с правильной тРНК при помощи аминоацил-тРНК-синтетазы (АРСаза) в два этапа.[8] На первом этапе используется энергия аденозинтрифосфорной кислоты (АТФ), синтезированной в другом месте при помощи АТФ-синтазы – фермента, содержащего миниатюрный роторный двигатель F1-ATPase.[9],[10],[11],[12] АТФ вступает в реакцию с аминокислотой и образует смешанный карбоксильно-фосфорный ангидрид.[13] На втором этапе аминоацильный остаток образует сложный эфир с 3’-гидроксилом рибозы, который расположен на концевом аденозине тРНК.[8],[13]
Коррекция неправильного продукта при помощи ферментов «двойного сита»
Тем не менее, этих этапов недостаточно, чтобы обеспечить необходимую высокую точность трансляции генетической информации (частота ошибок составляет от 1/2400 до 1/40 000). Фермент АРСаза также проводит коррекцию конечных продуктов, чтобы убедиться, что правильная аминокислота была связана с правильной тРНК. Одна из трудностей – как отличить друг от друга химически похожие аминокислоты. В частности, L-валин (Вал) и L-изолейцин (Иле) отличаются только одной метиленовой (CH2) группой. Лауреат двух Нобелевских премий Лайнус Полинг (Linus Pauling; 1901 — 1994) подсчитал, что поскольку группа CH2 имеет энергию гидрофобной связи всего около 4 кДж / моль, частота ошибок при замене Иле на Вал будет примерно один к пяти.[14] Таким образом, для обычного одноэтапного распознавания этих аминокислот термодинамически невозможно достичь уровня ошибок 1/3000, реально наблюдаемого у изолейцил-тРНК синтетазы (IleRS).[15],[16],[17],[18]
А ведь ошибка при замене Вал на Иле может быть биологически вредной или даже катастрофической. Даже единичная мутация Иле-Вал в ядре рибонуклеазы Т1 снижает стабильность этой молекулы, потому что «теряются правильные связи в боковой полипептидной цепи, когда белок находится в свёрнутой форме».[19] Такая мутация в гидрофобном ядре ингибитора химотрипсина 2 изменяет свободную энергию разворачивания белка (DDGU–F) в среднем на 5,0 ± 0,4 кДж / моль.[20] А единичная Иле-Вал мутация лизоцима человека приводит к меньшей устойчивости этого белка к денатурации (DDG от -1,5 до -5,0 кДж / моль).[21] Эта мутация также повышает предрасположенность к раку легких[22] и влияет на лекарственную устойчивость ВИЧ-1.[23]
Другая проблема, на которую указывал Полинг, заключается в том, что, хотя участок связывания фермента эффективно отсеивает стерически большие молекулы, каким образом он не пропускает молекулы меньшего размера (изостерические)?[14],[15]
Алан Фершт (Alan Fersht) был первым, кто в 1977 году предложил решение: механизм коррекции, или «двойного сита».[24] Первое грубое сито исключает активацию более крупных аминокислот, но позволяет активировать правильную специфическую аминокислоту, а также более мелкие. Затем второе мелкое сито отсеивает продукты более мелких (изостерических) аминокислот за счет их гидролизации (см. диаграмму ниже).
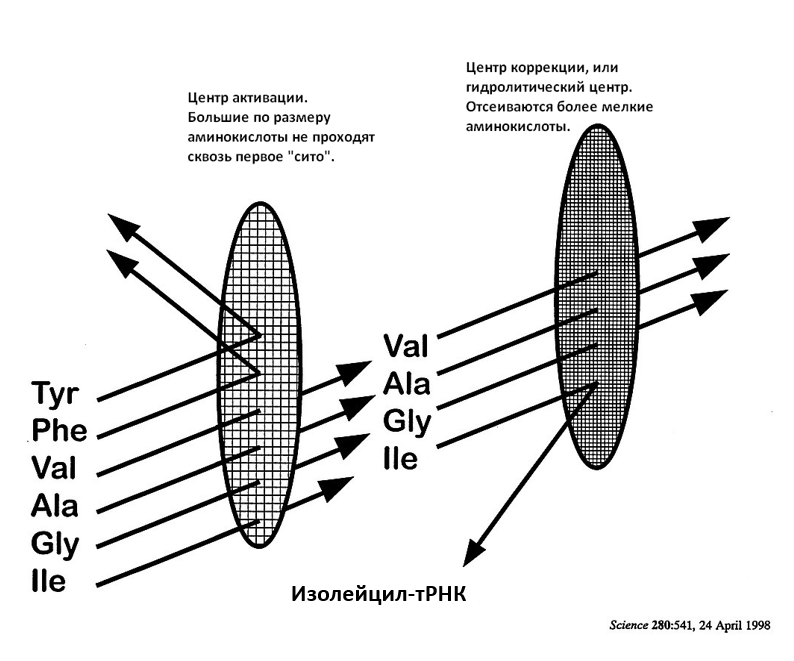
Рисунок 3. Механизм двойного сита, моделирующий функционирование изолейцил-тРНК-синтетазы. Гидролитическая коррекция снижает частоту ошибок при неправильной активации валина с ожидаемого значения между 1 к 10 и 1 к 100 — до 1 к 40 000 (согласно Ferscht[15]).
В 1998 году в работе Nureki et al. был продемонстрирован механизм двойного сита в молекуле IleRS. Был использован метод дифракции рентгеновских лучей для определения кристаллической структуры фермента IleRS у бактерии Thermus thermophilus, а также его комплексов с Иле и Вал. Фермент IleRS – это огромная L-образная молекула размером примерно 100 Å x 80 Å x 45 Å, принадлежащая к пространственной группе C2.[8]
В своём центре IleRS содержит укладку Россмана — характерный структурный мотив, участок, который связывает нуклеотиды. Первое «грубое сито» — это ямка в укладке Россмана с двумя специфическими последовательностями из четырех аминокислот, которые связывают АТФ. Ямка также связывает L-Ile в нижней части — углеводородные группы и группы NH3 + и COO– этой аминокислоты распознаются специальными стратегически расположенными аминокислотными остатками фермента. Этот центр способен отсеивать более крупные аминокислоты из-за стерических препятствий, включая L-лейцин, хотя он отличается от изолейцина (Ile) только размещением метильной группы на боковой цепи. Это так не похоже на нашу лабораторную органическую химию, где «лейцин и изолейцин особенно трудно разделить».[25]
Второе мелкое сито — это еще одна часть укладки Россмана, структурный домен Ins-2, который содержит еще одну глубокую ямку. При помощи дифракции рентгеновских лучей в этой ямке обнаруживается валин в комплексе L-валин-IleRS, но никогда не обнаруживается изолейцин в комплексе L-изолейцин-IleRS — ямка просто слишком мала. Здесь происходит коррекция неправильного продукта валина, но правильные продукты изолейцина защищены от гидролиза.
Nureki et al. продемонстрировали это, сконструировав мутантный IleRS, в котором отсутствовали 47 аминокислотных остатков, включая триптофан (Trp232) L-валин-специфического кармана.[8] Это полностью разрушило функцию коррекции. В другом эксперименте Nureki et al. подверг мутациям только две аминокислоты (заменив Thr243 и Asn250 на аланин) фермента IleRS кишечной палочки (E. сoli), что снова полностью разрушило функцию коррекции. Предыдущая работа показала, что даже единичная мутация (замена Tyr403 на фенилаланин (Phe)) значительно снижает коррекционную способность фермента IleRS кишечной палочки.[26]
Другие ферменты АРСаза также имеют функцию коррекции, включая молекулу ValRS, которая деацилирует неправильные продукты треонина.[27]
Эволюционное предубеждение
К сожалению, блестящая статья Nureki et al.[8] была испорчена тем, что авторы последовали за светским мейнстримом и преклонились перед идолом сегодняшнего дня — Нечестивой Троицей, состоящей из Времени, Случайности и Естественного Отбора. Вот что они пишут:
«…с эволюционной точки зрения интересно, что все ферменты, катализирующие центральные этапы биосинтеза и метаболизма Иле-Вал (изолейцин-валин), не различают или могут игнорировать разницу между двумя алифатическими аминокислотами, как это было показано для первого каталитического участка IleRS. Это открытие означает, что предполагаемый предковый фермент IleRS и ValRS мог на самом деле иметь аналогичную двойную специфичность для L-изолейцина и L-валина в системе первичного генетического кода».[28]
Конечно, хороший дизайнер часто использует похожее оборудование для изготовления аналогичных продуктов,[29] и это особенно имеет смысл, с учетом чрезвычайно похожего химического строения изолейцина и валина.[25] Так что заявление о предковом ферменте является просто сказкой без малейших доказательств, которая не может дать точного объяснения того, как такой центр коррекции (гидролитический центр) мог развиться в результате естественного отбора. Чтобы этот центр вообще мог работать, требуется огромное количество аминокислот, расположенных в строгой последовательности, и это демонстрирует отличительную черту дизайна — то, что биохимик Майкл Бихи в своей книге «Черный ящик Дарвина» назвал неуменьшаемой сложностью.[30] В нашем случае проблема стоит особенно остро — поскольку естественный отбор равен репродуктивному успеху, при плохой коррекции точное воспроизведение удачных признаков невозможно. Гораздо более вероятна катастрофическая ошибка.[29],[31],[32]
Ссылки и примечания
1. В качестве наглядной иллюстрации., см. Gitt, W., Dazzling Design in Miniature, Creation 20(1):6, 1997.
2. Fraser, C.M., et al. The minimal gene complement of Mycoplasma genitalium’, Science 270(5235):397–403, 1995; Perspective by A. Goffeau, Life with 482 genes, same issue, pp. 445–6.
3. Popper, K.R., Scientific reduction and the essential incompleteness of all science; in Ayala, F. and Dobzhansky, T., eds., Studies in the Philosophy of Biology, University of California Press, Berkeley, p. 270, 1974.
4. Хорошее описание — Denton, M., Evolution: a Theory in Crisis, Adler & Adler, Bethesda, Maryland, ch. 10, 1985.
5. Sarfati, J.D., Origin of life: the polymerization problem. CEN Tech. J. 12(3):281–284, 1998.
6. Mills, G.C. and Kenyon, D.H., The RNA World: A Critique, Origins and Design 17(1): 9–16, 1996.
7. Sarfati, J.D., Self-Replicating Enzymes? Journal of Creation. 11(1):4–6.
8. Nureki, O. and nine others, Enzyme structure with two catalytic sites for double-sieve selection of substrate, Science 280(5363):578–582, 1998.
9. Hiroyuki Noji et al., Direct observation of the rotation of F1-ATPase. Nature 386(6622):299–302, 1997; perspective by Block, S., Real engines of creation, same issue, pp. 217–219.
10. Boyer, P., The binding change mechanism for ATP synthesis—some probabilities and possibilities, Biochim. Biophys. Acta 1140:215–250, 1993. Return to text.
11. Abrahams, J.P. et al., Structure at 2.8 Å … resolution of F1-ATPase from bovine heart mitochondria. Nature 370(6491):621–628, 1994. Comment by Cross, R.L. Our primary source of ATP. Same issue, pp. 594–595. Return to text.
12. Sarfati, J.D., Дизайн в живых организмах (моторы: АТФ-синтазы), Journal of Creation. 12(1):3–5, 1998.
13. Karlson, P., (tr. Doering, C.H.), Introduction to modern biochemistry, 4th ed., Academic Press, NY, London, pp. 145–146, 113, 1975.
14. Pauling, L., in Festschrift Arthur Stoll, Birkhäuser Verlag, Basel, Switzerland, p. 597, 1958; cited in Nureki et al., Ref. 8.
15. Fersht, A.R., Sieves in sequence, Science 280(5363):541 (comment on Nureki et al., Ref. 8), 1998. Return to text.
16. Freist, W., Pardowitz, I. and Cramer, F., Isoleucyl-tRNA synthetase from baker’s yeast: multistep proofreading in discrimination between isoleucine and valine with modulated accuracy, a scheme for molecular recognition by energy dissipation, Biochemistry 24(24):7014–7023, 1995.
17. Loftfield, R.B., Biochem. J. 89:82–92, 1963; cited in Freist et al., Ref. 16.
18. Loftfield, R.B. and Vanderjagt, D., Biochem. J. 128:1353–1356, 1972; cited in Freist et al., Ref. 16.
19. Sneddon, S.F. and Tobias, D.J., 1992. The role of packing interactions in stabilizing folded proteins. Biochemistry 31(10):2842–2846.
20. Jackson, S.E. et al., 1993. Effect of cavity-creating mutations in the hydrophobic core of chymotrypsin inhibitor 2. Biochemistry 32(43):11259–11269.
21. Takano, K. et al., 1995. Contribution of hydrophobic residues to the stability of human lysozyme: caolimetric studies and x-ray structural analysis of the five isoleucine to valine mutants. J. Mol. Biol. 254:62–76.
22. Zhang, Z.Y. et al., 1996. Cancer Res. 56:3926; cited in Nureki et al., Ref. 8.
23. Farrash, M.A. et al., 1994. J. Virol. 68:233; cited in Nureki et al., Ref. 8.
24. Fersht, A.R., 1977. Enzyme Structure and Mechanism, Freeman, San Francisco, p. 283; cited in Fersht, Ref. 15.
25. Karlson, Ref. 13, p. 27.
26. Schmidt, E. and Schimmel, P., 1995. Residues in a Class I tRNA synthetase which determine selectivity of amino acid recognition in the context of tRNA. Biochemistry 34(35):11204–11210.
27. Eldred, E.W. and Schimmel, P., 1972. J. Biol. Chem. 247:2961; cited in Nureki et al., Ref. 8.
28. Nureki et al., Ref. 8, p. 581. [See p. 17 this journal.]
29. ReMine, W.J., The Biotic Message, St. Paul Science, St. Paul, MN, passim, 1993; see review by Batten, D., Journal of Creation. 11(3):292–298, 1997.
30. Behe, M.J., Darwin’s Black Box: The Biochemical Challenge to Evolution, The Free Press, New York, 1996; see review by Ury, T.H., Journal of Creation. 11(3):283–291, 1997. See also DiSilvestro, R., Rebuttals to common criticisms of the book Darwin’s Black Box (last updated 26 October 1999).
31. Denton, Ref. 4, ch. 11.
32. Jorde, L.B., Carey, J. C. and White, R.L., Medical Genetics, Mosby, St Louis, Missouri, 1995.
Если вам понравилась статья, поделитесь ею со своими друзьями в соц. сетях!










 При использовании материалов служения «Апологетика Библии» прямая ссылка на веб-сайт
При использовании материалов служения «Апологетика Библии» прямая ссылка на веб-сайт